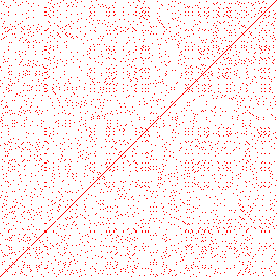
Dot-Plot-Analyse
Home l Extractor l CoordCalc l Viewer l Begriffe
![]()
Download Viewer (163k), exe
Download Delphi-Sourcecode (227k), zip
[Läuft nur unter Win 98, NT & XP]
![]()
![]()
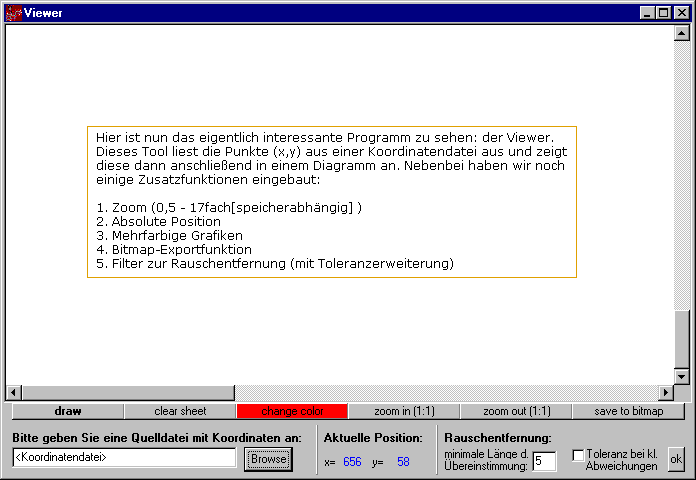
Zur grafischen Darstellung liest das Programm nach einem Klick auf "draw" zuerst die Maximalwerte für die Anpassung der Fenstergröße und dann die Koordinaten aus der unter Quelldatei angegebenen *.cor-Datei nacheinander ein. Für jede Koordinate wird ein Punkt in der aktuellen Farbe gesetzt.
1. Zoom
Unser Tool ist in der Lage, eine Grafik 0,5 - 17fach (bei 2000*2000 Bildpunkten; speicherabhängig) zu vergrößern. Dies ist über die Picture-Stretch-Funktion der Image-Komponente realisiert.
2. Absolute Position
Im unteren mittleren Bildschirmbereich wird stets die aktuelle Position im Koordinatensystem ausgegeben, damit man Übereinstimmungen der Sequenz auch in der Proteinkette später wieder auffinden kann.
3. Mehrfarbige Grafiken
Die Zeichenfarbe ist über "change color" veränderbar; die Löschung der alten Anzeige geschieht erst über "clear sheet". Sollte die Zeichenroutine einen bereits farbigen Punkt vorfinden, d.h. die beiden Grafiken überlagern sich, so werden die Farbwerte addiert (Mischung -> blau und rot wird zu violett).
4. Bitmap-Exportfunktion
Natürlich ist eine Abspeicherung der Grafik in eine Bitmap-Datei möglich, z.B. zur Verwendung in Office-Dokumenten oder anderen externen Applikationen.
5. Filter zur Rauschentfernung (mit Toleranzerweiterung)
Um längere Übereinstimmungen in den Ketten besser erkennen zu können, werden durch diesen Filter alleinstehende Punkte, die auf keiner Diagonalen liegen (d.h. nicht zu einem längeren identischen Sequenzabschnitt gehören), herausgeschnitten.
Die Länge der minimal zu beachtenden Diagonalenlänge (Übereinstimmung) kann frei festgelegt werden, Vorgabewert ist 5.
Nun kann es vorkommen, dass (z.B. durch Mutation) in einer längeren Übereinstimmung eine kleine Abweichung auftritt. Damit nun nicht der ganze Abschnitt verworfen wird, werden folgenden "Fehler" toleriert:
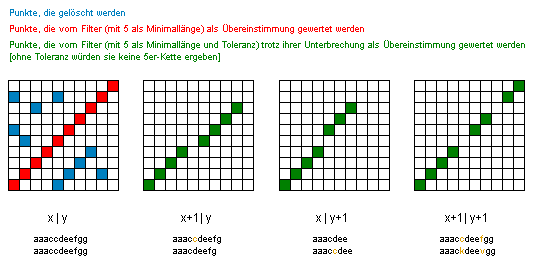
Das gefilterte Ergebnis kann dann in eine neue Koordinatendatei (*.cor) abgespeichert werden. Für den Vergleich von "Helicobacter pylori, cag7" mit sich selbst sieht diese dann so aus [Ausschnitt]:
![]()
Beispiele
1. Vergleich "Helicobacter pylori, Cag 7" mit sich selbst


ungefiltert
gefiltert (Minimallänge: 5, mit Toleranz)
deutlich erkennbar: Symmetrie, da das Bakterium mit sich selbst verglichen wurde
2. Vergleich "Helicobacter pylori, Cag 7" mit einer Zufallskette
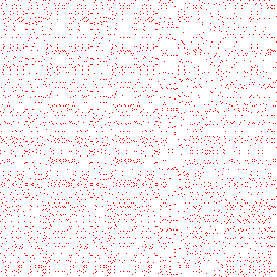

ungefiltert
gefiltert (Minimallänge: 5, mit Toleranz)
deutlich erkennbar: fast keine Übereinstimmungen, da es keinen Zusammenhang zwischen der zufälligen Kette und dem Bakterium gibt
3. Vergleich "Prion-Protein" mit sich selbst


ungefiltert
gefiltert (Minimallänge: 5, mit Toleranz)
deutlich erkennbar: Symmetrie, da das Protein mit sich selbst verglichen wurde
4. Vergleich "Humaner Grippe-Virus: Influenza A virus (A/Indiana/28170/99(H3N2))" mit
"Humaner Grippe-Virus: Influenza A virus (A/Utah/20997/99(H3N2))"
hemagglutinin [Influenza A virus (A/Indiana/28170/99(H3N2))]
hemagglutinin [Influenza A virus (A/Utah/20997/99(H3N2))]
![]()