Dot-Plot-Analyse |
![]()
Download Extractor (157k), exe
Nachdem man eine Quelldatei angegeben hat, wird automatisch eine Zieldatei (ASCII-Textformat) mit der Endung *.ptn vorgeschlagen. Diese enthält später nur noch die sequenzielle Zeichenfolge der Bakterie.
Nachdem wir die Proteinkette aus der HTML-Datei des "Helicobacter pylori, cag7" extrahiert hatten, erhielten wir eine Datei in folgendem Format (*.ptn):
Download Delphi-Sourcecode (214k), zip
[Läuft nur unter Win 98, NT & XP]
![]()
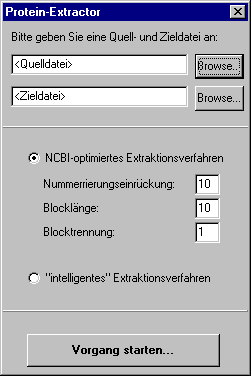
Der Nutzer kann sich zwischen zwei Suchverfahren entscheiden, nach denen die Datei gefiltert werden soll:
1. NCBI-Verfahren
Wir haben festgestellt, dass die Texte aus der NCBI-Datenbank immer gleich aufgebaut sind. Zuerst kommt ein Kopf mit Angaben, dann folgt das Wort ORIGIN und danach die Kette in einem festen Blocksatz (10 Zeichen Nummerierungseinrückung gefolgt von 6 10er-Blöcken der Bakteriensequenz, alles jeweils von einem Leerzeichen getrennt). Als Abschluss wird // genutzt. So konnten wir speziell für diese Quelldateien ein Extraktionsverfahren entwickeln.
2."intelligentes" Suchverfahren
Da unser Programm auch mit leicht anders aufgebauten Quelldateien noch zurecht kommen sollte, programmierten wir ein weiteres "intelligentes" Verfahren. Es sucht ebenfalls zuerst nach dem Indentifikationswort ORIGIN, löscht dann allerdings nicht nach Blockgröße, sondern entfernt überflüssige Leerzeichen, Zahlen usw., bis nur noch die Buchstabenkette zurück bleibt. Auch hier gilt // als Abschlusszeichen.
Im nächsten Schritt entstand ein weiteres kleines Teilprogramm, das zufällige Ketten erstellte, da wir auch hier Vergleiche mit Bakterien anstellen wollten...
--> mehr![]()
![]()