Dot-Plot-Analyse |
![]()
Download CoordCalc (161k), exe
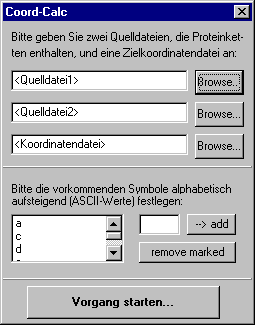
Unser zweites Teilprogramm dient der Umrechung von Übereinstimmungen aus zwei Proteinketten (*.ptn-Dateien) in Koordinaten (*.cor-Datei). Nachdem man zwei Proteindateien angegeben hat, wird automatisch ein Name für die Zieldatei vorgeschlagen, welche dann die Koordinaten enthält (diese Datei kann mitunter sehr umfangreich sein).
Nachdem die Proteinketten verglichen wurden, schreibt das Programm die Koordiantendatei in folgendem Format (*.cor)
Download Delphi-Sourcecode (220k), zip
[Läuft nur unter Win 98, NT & XP]
![]()
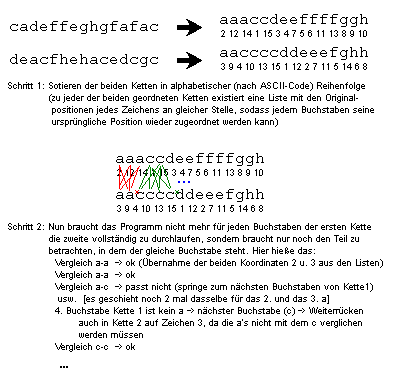
Da es ineffektiv gewesen wäre, jedes Symbol der ersten Kette mit jedem Symbol der zweiten Kette zu vergleichen und bei Übereinstimmung eine Koordinate aufzunehmen (das hätte nämlich eine quadratische Steigerung des Zeitaufwandes zur Kettenlänge zur Folge), mussten wir uns einen effektiveren Algorithmus überlegen, dessen Zeitverbrauch nur linear mit der Kettenlänge steigt. Vereinfacht ausgedrückt arbeitet dieser folgendermaßen:
Nachdem man die vorkommenden Zeichen definiert hat (insofern a-y nicht zutreffend sind), berechnet ein Klick auf "Vorgang starten" die Koordinaten und schreibt diese auf die Angabe von Breite und Höhe des Koordinatensystems (Länge der ersten und der zweiten Proteinkette) folgend in die angegebene Zieldatei (*.cor).![]()
[Ausschnitt des Vergleichs von "Helicobacter pylori, cag7" mit sich selbst]:![]()